TIANSeq Single-Index Adapter (Illumina)
Eegeschaften
■ Praktesch: SetA a SetB béid liwweren 24 eenzegen Index, dee fräi no Bedierfnesser ausgewielt ka ginn.
■ Einfach ze benotzen: De Kit ass mat Adapter Verdünnungsbuffer ausgestatt, deen eng staark Stabilitéit huet an direkt fir d'Verdünnung vun der Adapterléisung benotzt ka ginn.
■ Qualitéitskontroll: Strikt Qualitéitskontroll a funktionell Verifizéierung tëscht Chargen garantéieren d'Genauegkeet vun der Indexsequenz.
Uwendungen
1. Dëst Produkt gëtt fir de Bau vun DNA a RNA Bibliothéike fir Illumina High-Throughput Sequencing Plattform an der Uwendung vun der nächster Generatioun Sequencing (NGS) benotzt.
2. Spezifesch Uwendungen vum Produkt enthalen ganz Exon Sequencing, geziilte Sequencing, RNA- Seq, ChIP-Seq, geriicht Sequencing a Ganz Genom Sequencing.
3. Et sollt bemierkt datt dëst Produkt net gëeegent ass fir methyléierungsbedéngte Sequencing.
Adapter Sequenz Informatioun
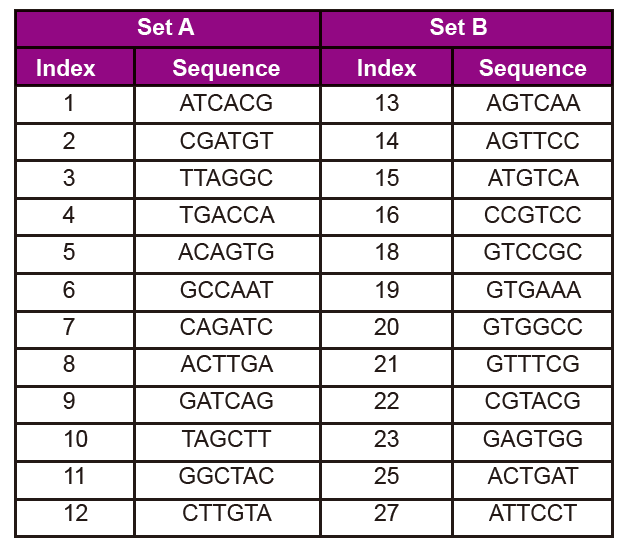
D'Adaptersequenz enthält déi folgend Informatioun:
1. Universal Sequenz
5'-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCT
ACACGACGCTCTTCCGATCT-3 '
2. Index Inklusive Sequenz
5'-GATCGGAAGAGACACACGTCTGAACTCCAGTCAC [index1-27] ATCTCGTATGCCGTCTTCTGCTTG-3 '
3. Indexnummer a Sequenz
All d'Produkter kënne fir ODM/OEM personaliséiert ginn. Fir Detailer,klickt w.e.g. Customized Service (ODM/OEM)
De Moment ass High-Throughput Sequencing Technologie haaptsächlech baséiert op der nächster Generatioun Sequencing Technologie. Wéi d'Lieselängt vun der nächster Generatioun Sequencing Technologie limitéiert ass, musse mir déi ganz Längt Sequenz a kleng Fragmentbibliothéike briechen fir ze sequenzéieren. Geméiss de Bedierfnesser vu verschiddene Sequencing Experimenter, wiele mir normalerweis Single-End Sequencing oder Double-End Sequencing. De Moment sinn d'DNA Fragmenter vun der nächster Generatioun Sequenzenbibliothéik allgemeng am Beräich vun 200-800 BP verdeelt.

a) DNA ass schlecht a Qualitéit an enthält Inhibitoren. Benotzt héichqualitativ DNA Proben fir Inhibitioun vun der Enzymaktivitéit ze vermeiden.
b) De Betrag vun der DNA Probe ass net genuch wann Dir PCR-gratis Method benotzt fir d'DNA Bibliothéik ze bauen. Wann den Input vun der fragmentéierter DNA méi wéi 50 ng ass, kann PCR-gratis Workflow selektiv wärend dem Bibliothéikbauprozess duerchgefouert ginn. Wann d'Kopiezuel vun der Bibliothéik ze niddreg ass fir direkt sequenzéiert ze ginn, kann d'DNA -Bibliothéik duerch PCR verstäerkt ginn no der Adapterligatioun.
c) RNA Kontaminatioun féiert zu ongenau initial DNA Quantifizéierung RNA Kontaminatioun kann am Botzprozess vu genomescher DNA existéieren, wat zu enger ongenauer DNA Quantifizéierung an net genuch DNA Luede wärend der Bibliothéikskonstruktioun féiere kann. RNA ka geläscht ginn andeems se mat RNase behandelt ginn.
A-1 an
a) Kleng Fragmenter (60 bp-120 bp) erschéngt Kleng Fragmenter si meeschtens Adapterfragmenter oder Dimeren, déi duerch Adapter geformt ginn. Reinigung mat Agencourt AMPure XP magnetesche Pärelen kënnen dës Adapterfragmenter effektiv ewechhuelen an d'Sequenzéierungsqualitéit garantéieren.
b) Grouss Fragmenter erschéngen an der Bibliothéik no PCR Verstäerkung D'Gréisst vum Bibliothéik DNA Fragment wäert ëm 120 BP eropgoen nodeems den Adapter ligéiert ass. Wann d'DNA Fragment ëm méi wéi 120 BP no der Adapterligatioun eropgeet, kann et duerch eng anormal Fragmentamplifikatioun vun enger exzessiver PCR Amplifikatioun verursaacht ginn. D'Zuel vun de PCR Zyklen ze reduzéieren kann d'Situatioun vermeiden.
c) Abnormal Gréisst vu Bibliothéik DNA Fragmenter no Adapterligatioun D'Längt vum Adapter an dësem Kit ass 60 BP. Wann déi zwee Enden vum Fragment op d'Adaptere ligéiert sinn, wäert d'Längt nëmmen ëm 120 BP eropgoen. Wann Dir en aneren Adapter benotzt wéi dee vun dësem Kit, kontaktéiert w.e.g. den Zouliwwerer fir relevant Informatioun ze liwweren wéi d'Längt vum Adapter. Gitt sécher datt den Experiment Workflow an d'Operatioun de Schrëtt verfollegen, déi am Handbuch beschriwwe sinn.
d) Abnormal DNA Fragmentgréisst virun der Adapterligatioun De Grond fir dëse Problem kann duerch falsch Reaktiounsbedéngungen wärend der DNA Fragmentéierung verursaacht ginn. Verschidde Reaktiounszeeche solle fir verschidden DNA Input benotzt ginn. Wann d'DNA Input méi wéi 10 ng ass, empfeelen mir d'Reaktiounszäit vun 12 min als Startzäit fir Optimiséierung ze wielen, an d'Fragmentgréisst, déi zu dëser Zäit produzéiert gëtt, ass haaptsächlech am Beräich vun 300-500 BP. D'Benotzer kënnen d'Längt vun DNA Fragmenter fir 2-4 min jee no hiren eegenen Ufuerderunge erhéijen oder erofgoen fir d'DNA Fragmenter mat der erfuerderlecher Gréisst ze optimiséieren.
A-2 an
a) Fragmentatiounszäit ass net optimiséiert Wann d'fragmentéiert DNA ze kleng oder ze grouss ass, kuckt w.e.g. d'Richtlinne fir d'Fragmentatiounszäitauswiel an der Instruktioun zur Verfügung fir d'Reaktiounszäit ze bestëmmen, a benotzt dësen Zäitpunkt als Kontroll, setzt zousätzlech e Reaktiounssystem fir 3 min ze verlängeren oder ze verkierzen fir méi präzis Upassung vun der Fragmentatiounszäit ze maachen.
A-3 an
Abnormal Gréisst Verdeelung vun DNA no Fragmentéierungsbehandlung
a) Falsch Ausdiermethod vum Fragmentéierungsreagens, oder de Reagens ass net komplett gemëscht nom Téi. Thaw de 5 × Fragmentatioun Enzym Mix Reagens op Äis. Eemol enttäuscht, mëscht de Reagens gläichméisseg andeems de Buedem vum Rouer sanft flitt. Wirbel de Reagens net!
b) D'DNA Input Probe enthält EDTA oder aner Schuedstoffer Ofbau vu Salzionen a Chelatmëttelen am DNA Reinigungsstuf ass besonnesch wichteg fir den Erfolleg vum Experiment. Wann d'DNA an 1 × TE opgeléist gëtt, benotzt d'Method, déi an der Instruktioun gëtt fir Fragmentéierung ze maachen. Wann d'EDTA Konzentratioun an der Léisung onsécher ass, ass et recommandéiert d'DNA ze purifizéieren an se an deioniséiertem Waasser fir eng spéider Reaktioun ze léisen.
c) Ongenau initial DNA Quantifizéierung D'Gréisst vun der fragmentéierter DNA ass enk mat der Quantitéit vum DNA Input verbonnen. Virun der Fragmentéierungsbehandlung ass eng korrekt Quantifizéierung vun DNA mat Qubit, Picogreen an aner Methoden essentiell fir d'exakt Quantitéit vun DNA am Reaktiounssystem ze bestëmmen.
d) D'Virbereedung vum Reaktiounssystem follegt d'Instruktioune net D'Virbereedung vum fragmentéierten Reaktiounssystem muss strikt no den Instruktiounen op Äis ausgefouert ginn. Fir dee beschte Effekt ze garantéieren, sollten all Reaktiounskomponenten op Äis geluecht ginn an d'Virbereedung vum Reaktiounssystem soll nom komplette Ofkillung duerchgefouert ginn. Nodeems d'Virbereedung fäerdeg ass, flitt w.e.g. oder pipet fir grëndlech ze mëschen. Wirbel net!
1. Ongerechteg Mëschungsmethod (Wirbel, gewaltsam Schwéngung, asw.) Verursaacht eng anormal Verdeelung vu Bibliothéikfragmenter (wéi an der folgender Figur gewisen), sou beaflosst d'Qualitéit vun der Bibliothéik. Dofir, wann Dir d'Fragmentéierung Mix Reaktiounsléisung virbereet, pipéiert w.e.g. sanft op an erof fir ze mëschen, oder benotzt de Fanger fir fléissend a gläichméisseg ze mëschen. Sidd virsiichteg net mam Wirbel ze vermëschen.

2. Héich Rengheet DNA muss fir de Bau vun der Bibliothéik benotzt ginn
■ Gutt DNA Integritéit: D'Elektrophorese Band ass méi wéi 30 kb, ouni Schwanz
■ OD260/230:> 1.5
■ OD260/280: 1,7-1,9
3. DNA Input Betrag muss korrekt sinn Et gëtt virgeschlo Qubit a PicoGreen Methoden ze benotzen fir DNA ze quantifizéieren, anstatt Nanodrop.
4. Den Inhalt vun EDTA an der DNA Léisung muss bestëmmt ginn EDTA huet e groussen Afloss op d'Fragmentéierungsreaktioun. Wann den Inhalt vun EDTA héich ass, muss d'DNA Reinigung virum nächste Test ausgefouert ginn.
5. D'Fragmentéierungsreaktiounsléisung muss op Äis virbereet ginn De Fragmentéierungsprozess ass empfindlech op d'Reaktiounstemperatur an d'Zäit (besonnesch nodeems en Enhancer bäigefüügt gouf). Fir d'Genauegkeet vun der Reaktiounszäit ze garantéieren, bereet w.e.g. Reaktiounssystem op Äis vir.
6. D'Fragmentéierungsreaktiounszäit muss richteg sinn D'Reaktiounszäit vum Fragmentéierungsstuf beaflosst direkt d'Gréisst vun de Fragmentprodukter, sou beaflosst d'Gréisstverdeelung vun DNA Fragmenter an der Bibliothéik.
1. Wéi eng Probe ass fir dëse Kit uwendbar?
Déi uwendbar Probe -Aart vun dësem Kit kann total RNA oder gereinegt mRNA mat gudder RNA Integritéit sinn. Wann total RNA benotzt gëtt fir d'Bibliothéik ze bauen, ass et recommandéiert de rRNA Erschöpfungskit (Cat#4992363/4992364/4992391) ze benotzen fir d'éischt rRNA ze läschen.
2. Kann FFPE Proben benotzt gi fir Bibliothéik mat dësem Kit ze bauen?
D'MRNA an FFPE Proben ginn zu engem gewësse Mooss degradéiert, mat relativ aarmer Integritéit. Wann Dir dëse Kit fir d'Bibliothéik Konstruktioun benotzt, gëtt et recommandéiert d'Fragmentéierung Zäit ze optimiséieren (d'Fragmentéierung Zäit verkierzen oder d'Fragmentéierung net ausféieren).
3. Mat der Gréisst Auswiel Schrëtt am Produktmanual geliwwert, wat kéint dozou féieren datt de agebaute Segment eng kleng Ofwäichung erschéngt?
D'Gréisstauswiel soll a strikt Aklang mat der Gréisst Auswiel Schrëtt an dësem Produktmanual duerchgefouert ginn. Wann et Ofwäichung ass, kann de Grond sinn datt d'Magnéitpärelen net op Raumtemperatur ausgeglach sinn oder net voll gemëscht sinn, d'Pipette net richteg ass oder d'Flëssegkeet bleift am Tipp. Et ass recommandéiert d'Tipps mat gerénger Adsorptioun fir den Experiment ze benotzen.
4. Auswiel vun Adapteren am Bibliothéikskonstruktioun
De Bibliothéik Konstruktiounskit enthält keen Adapterreagens, an et ass recommandéiert dëse Kit zesumme mam TIANSeq Single-Index Adapter (Illumina) (4992641/4992642/4992378) ze benotzen.
5. QC vun der Bibliothéik
Bibliothéik quantitativer Detektioun: Qubit a qPCR gi benotzt fir d'Masskonzentratioun an d'Molekonzentratioun vun der Bibliothéik respektiv ze bestëmmen. D'Operatioun ass strikt am Aklang mat dem Produktmanual. D'Konzentratioun vun der Bibliothéik entsprécht allgemeng den Ufuerderunge vum NGS Sequencing. Detektioun vu Bibliothéikverdeelungsbereich: Benotzt Agilent 2100 Bioanalyzer fir d'Bibliothéikverdeelungsbereich z'entdecken.
6. Auswiel vun der Verstärkungszyklusnummer
Geméiss den Instruktiounen ass d'Zuel vu PCR Zyklen 6-12, an d'Zuel vun de PCR Zyklen déi gebraucht solle gewielt ginn no dem Probe Input. An Héichausbezuelungsbibliothéike geschitt Iwwerverstäerkung normalerweis a variabelen Grad, wat sech duerch e bësse méi groussen Héichpunkt no der Peak vum Zilberäich bei der Detektioun vum Agilent 2100 Bioanalyzer manifestéiert, oder déi festgestallt Konzentratioun vu Qubit ass méi niddereg wéi déi vum qPCR. Mëll iwwer Verstäerkung ass en normale Phänomen, dat keng Afloss op d'Bibliothéiksequenzéierung an déi spéider Dateanalyse huet.
7. Spikes erschéngen am Detektiounsprofil vum Agilent 2100 Bioanalyzer
D'Erscheinung vu Spikes bei der Agilent 2100 Bioanalyzer Detektioun ass wéinst der ongläicher Fragmentéierung vu Proben, wou et méi Fragmenter a bestëmmter Gréisst ginn, an dëst gëtt méi offensichtlech no PCR Beräicherung. An dësem Fall gëtt virgeschloen d'Gréisstauswiel net auszeféieren, dh de Fragmentéierungszoustand op 94 ° C fir 15 min incubéiert ze setzen, wou d'Fragmentverdeelung kleng a konzentréiert ass, an d'Homogenitéit ka verbessert ginn.
Produkter Kategorien
FIRWAT WËLLT EIS
Zënter hirer Grënnung huet eis Fabréck éischt Weltklass Produkter entwéckelt mam Prinzip ze halen
vun der Qualitéit als éischt. Eis Produkter hunn en exzellente Ruff an der Industrie gewonnen a wäertvoll Vertrauen ënner neien an alen Clienten ..








